Fagos para la detección de autoanticuerpos
Las respuestas inmunitarias dirigidas a las autoproteínas (autoantígenos) pueden conducir a una variedad de enfermedades autoinmunes. La identificación de estos antígenos es importante tanto por razones diagnósticas como terapéuticas. Sin embargo, los enfoques actuales para caracterizar tales entidades proteicas, en la mayoría de los casos, solo han tenido un éxito limitado. Recientemente, se ha realizado una representación sintética del proteoma humano completo, mediante una librería basada en el bacteriófago T7, el que es capaz de desplegar la totalidad de las secuencias del peptidoma humano. Su empleo ha permitido descubrir diversos autoantígenos. En simple, esto sucede por el desarrollo de una metodología de inmunoprecipitación de fagos a los que se les ha unido una inmunoglobulina IgG autorreactiva contenidos en líquido cefalorraquídeo de individuos con síndromes neurológicos paraneoplásicos. De esta forma, con el uso de esta herramienta molecular será posible identificar autoanticuerpos conocidos y aquellos no reportados anteriormente, con la finalidad de aplicar terapias específicamente dirigidas a las complicadas patologías autoinmunes.
Uso del Fago T7
El Premio Nobel de Química 2018 fue otorgado a tres científicos: Frances Arnold (California Institute of Technology), George Smith (Universidad de Missouri) y Gregory Winter (MRC Laboratory of Molecular Biology, Cambridge, Reino Unido), por su trabajo sobre la "evolución dirigida" de las proteínas. Estos investigadores fueron pioneros en los métodos in vitro basados en una característica y un principio de evolución -la diversidad genética y la selección- para diseñar y mejorar las moléculas de elección. Tanto Smith como Winter usaron bacteriófagos (fagos) para identificar péptidos y anticuerpos que ahora se utilizan en el diagnóstico y tratamiento de enfermedades, incluyendo enfermedades autoinmunes, infecciones y cáncer.
Smith describió por primera vez la tecnología de visualización de fagos en 1985, estimulado por su descubrimiento de que los fagos filamentosos podían ser diseñados para mostrar secuencias de aminoácidos exógenos en la punta de las proteínas de la capa proteica (cápside) sin destruir la infectividad del fago o la propagación en su huésped, Escherichia coli1 (figura 1A). El poder de la visualización del fago es proporcionado por la conexión física entre la proteína de cubierta modificada y el gen que la codifica. Cada clon de fagos muestra sólo una secuencia peptídica, pero una biblioteca de fagos muestra miles de millones de péptidos diferentes. Esta librería puede ser examinada a través de un proceso conocido como "selección de afinidad", para identificar qué fago específico expresa un polipéptido con una propiedad de unión específica.
En 1990, Winter y sus colegas reportaron la exhibición de fragmentos de anticuerpos humanos tales como fragmentos variables de cadena única (scFv) en fagos filamentosos (Nature 1990;348:552-554). Los fragmentos de anticuerpos seleccionados de las bibliotecas de exhibición de fagos filamentosos han tenido el mayor éxito en la traducción a la clínica, en comparación con otras plataformas de fagos. El adalimumab se une al factor de necrosis tumoral, y en 2002 fue la primera molécula seleccionada de exhibición de fagos aprobada por la Food and Drug Administration para tratar enfermedades humanas. Se utiliza para tratar enfermedades autoinmunes inflamatorias, incluyendo la artritis reumatoide. El uso de las bibliotecas de scFv también permitió la selección de anticuerpos contra patógenos, lo que condujo al desarrollo de raxibacumab, que se une al antígeno protector de Bacillus anthracis y fue aprobado en 2012 para la prevención y el tratamiento del ántrax inhalado.
Otro tipo de fago, el T7, utilizado ampliamente en investigación y, en menor medida, en aplicaciones clínicas. Los fagos T7 difieren de los filamentosos en su estructura general, replicación lítica y núcleo de ADN lineal de doble cadena (figura 1A). Los fagos T7 crecen más rápidamente y tienen mayor estabilidad del genoma y capacidad para contener insertos de ADN grandes (>3.000 pb) que los fagos filamentosos. En 2011, Larman y sus compañeros de trabajo (Nat Biotechnol. 2011 May 22;29(6):535-41) reportaron la creación de una biblioteca de exhibición de polipéptidos T7 derivados de un proteoma humano sintético. La biblioteca se construyó con "azulejos" (cada uno compuesto de 36 aminoácidos) con residuos superpuestos. Cada biblioteca contenía 413.000 péptidos diferentes, suficientes para cubrir el proteoma humano (es decir, todas las proteínas previstas por la secuencia de codificación del genoma humano). Las bibliotecas fueron inmunoprecipitadas con anticuerpos de pacientes para la recuperación del autoantígeno, y los fagos seleccionados fueron analizados por secuenciación de ADN de alto rendimiento. Por lo tanto, todo el proteoma humano puede ser examinado, con sólo una o dos rondas de selección de afinidad (figura 1B). Los enfoques previos a la identificación de los autoantígenos incluían el cribado de bibliotecas de expresión de ADN complementarias con autoantisuero del paciente, un proceso ineficiente y que consumía mucho tiempo.
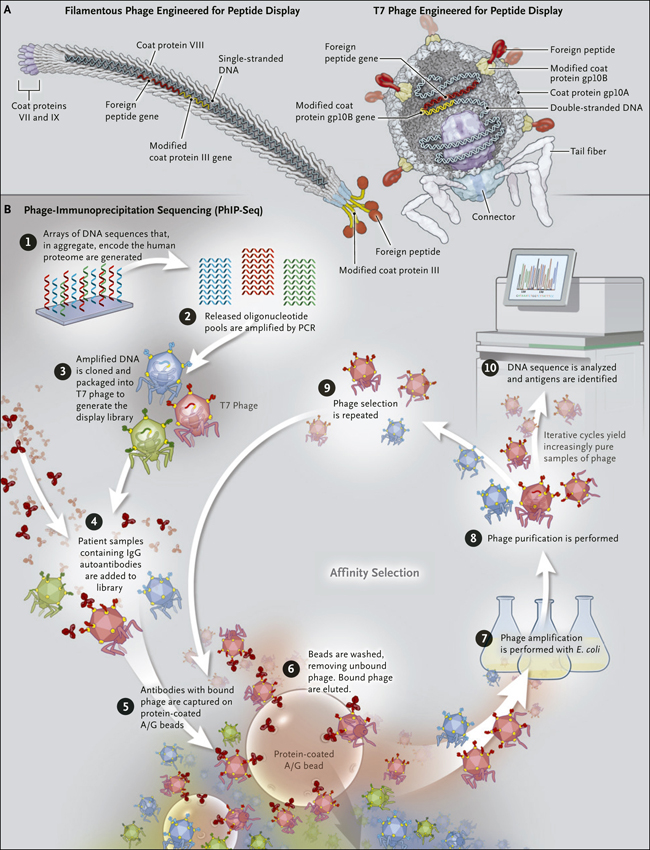
Figura 1. Selección por afinidad de secuencias de unión a autoanticuerpos por despliegue de fagos.
El panel A muestra una comparación de fagos filamentosos y viriones de fagos T7. Los fagos filamentosos son virus de ADN circular de cadena simple y que han sido diseñados para mostrar secuencias de aminoácidos extraños en la proteína de cubierta III y la proteína de cubierta VIII. Los fagos T7 se distinguen por su cabeza icosaédrica (cápside) y su genoma lineal de ADN de doble cadena. Han sido diseñados para mostrar polipéptidos en la proteína de cubierta gp10B. El panel B muestra la secuencia de inmunoprecipitación (PhIP-Seq), el método utilizado en el estudio de Mandel-Brehm y colaboradores (N Engl J Med 2019;381:47-54). Se utilizó una biblioteca de visualización de fase T7 para identificar la causa de una encefalitis paraneoplásica, una enfermedad que se presume autoinmune. Los autores crearon esta biblioteca a partir de ADN que codifica el proteoma humano. Las secuencias de codificación de péptidos se sintetizaron como grupos de 49 oligonucleótidos de aminoácidos en matrices de ADN (paso 1), y los grupos de oligonucleótidos liberados se amplificaron mediante la reacción en cadena de la polimerasa (PCR) (paso 2). Los grupos de ADN fueron clonados en ADN codificante para la proteína de cubierta gp10B del fago T7 para crear la biblioteca (paso 3), compuesta de fagos, cada uno de los cuales expresaba un solo péptido. La biblioteca se seleccionó por medio de la evaluación de muestras de pacientes (suero, así como líquido cefalorraquídeo que presumiblemente contenía autoanticuerpos IgG) (paso 4). Se capturaron anticuerpos ligados a fagos o se inmunoprecipitaron en perlas de agarosa recubiertas con una proteína que se une a IgG (paso 5). Las perlas se lavaron, liberando las superficies de las perlas de fagos no ligados o ligados de manera laxa, y luego se eluyeron los fagos ligados (paso 6). Los fagos eludidos fueron amplificados en Escherichia coli (paso 7) y posteriormente purificados (paso 8). Los fagos amplificados fueron usados en otra ronda de selección (paso 9). A continuación, se obtuvo la secuencia de ADN del fago de salida, lo que permitió la identificación del antígeno (paso 10).
Recientemente, Mandel-Brehm y sus colaboradores (N Engl J Med 2019;381:47-54) reportan el uso de una biblioteca de proteomas humanos T7 (figura 1B), similar a la desarrollada por Larman y sus colegas para descubrir autoanticuerpos contra la proteína tipo Kelch 11 en una serie de 13 hombres, 11 de los cuales tenían un diagnóstico de encefalitis paraneoplásica (autoinmune) asociada con el seminoma y 2 de los cuales tenían encefalitis autoinmune asociada con microlitiasis testicular y la atrofia testicular, una afección supuestamente premaligna. Esta biblioteca contenía más de 700.000 péptidos diferentes (cada uno de los cuales constaba de 49 aminoácidos). El motivo de kelch es una secuencia peptídica de aproximadamente 50 aminoácidos. Aunque el papel de la proteína tipo Kelch 11 en la enfermedad autoinmune y el cáncer no está claro, los autoanticuerpos para esta proteína representan un biomarcador candidato para la enfermedad.
Las enfermedades neurológicas paraneoplásicas, incluyendo la encefalitis paraneoplásica asociada al seminoma, ocurren en menos del 1% de las personas con cáncer y son mediadas por reacciones inmunológicas contra los autoantígenos compartidos por las células cancerosas y el sistema nervioso. Los autoanticuerpos (IgG) contra el antígeno paraneoplásico Ma2 han sido identificados previamente: los 13 pacientes del estudio de Mandel-Brehm y cols. resultaron negativos para la IgG Ma2. En 8 de estos hombres, el síndrome paraneoplásico precedió al diagnóstico de cáncer testicular. Por lo tanto, el cribado de pacientes varones con encefalitis para detectar autoanticuerpos de la proteína tipo Kelch 11 podría facilitar el diagnóstico, predecir el cáncer testicular y quizás contribuir de otra manera al control de la enfermedad.
El estudio de Mandel-Brehm y sus colegas demuestra muy bien el poder del despliegue de fagos para identificar la causa de una enfermedad autoinmune. Sin embargo, la tecnología es versátil y puede servir para otros objetivos, como el diseño de nuevos y mejores fármacos biológicos. Un buen ejemplo es su uso en la inmunoterapia, en particular en la terapia con células T receptoras de antígenos quiméricos (CAR-T). Las bibliotecas de receptores de células T humanas fueron creadas para identificar receptores "evolucionados" y nuevos que tienen una afinidad extremadamente alta con los antígenos humanos asociados al tumor. A partir de esto, fue posible modificar las células T de un paciente con los receptores de células T evolucionados para que puedan reconocer y destruir mejor sus células cancerosas. Con este ejemplo en mente, el futuro parece prometedor para la aplicación de la evolución molecular que implica la exhibición de fagos para el descubrimiento de fármacos.
Fuente bibliográfica
Phage Display to Detect and Identify Autoantibodies in Disease
Susan Deutscher, Ph.D.
Harry S. Truman Memorial Veterans’ Hospital Research Service and the Department of Biochemistry, University of Missouri — both in Columbia.
DOI: 10.1056/NEJMcibr1903249
Temas Relacionados