Detección de virus, más allá de CRISPR
La gran mayoría de los patógenos que circulan en el mundo no se detectan, lo que socava la atención de pacientes y obstaculiza la preparación y respuesta ante brotes infecciosos. Para hacer posible la vigilancia rutinaria y las aplicaciones de un diagnóstico integral, se necesitan tecnologías de detección que puedan ampliarse para analizar múltiples muestras y al mismo tiempo analizar muchos patógenos. Este anhelo clínico podría resolverse con una plataforma basada en reacciones combinadas para la evaluación de múltiples ácidos nucleicos (tecnología al que los investigadores han bautizado como CARMEN, por Combinatorial Arrayed Reactions for Multiplexed Evaluation of Nucleic acids). Utilizando este enfoque se logró analizar simultáneamente 169 virus asociados a humanos el agente etiológico de la COVID-19, el SARS-CoV-2. La tecnología además permite la subtipificación exhaustiva de las cepas de influenza y la identificación de docenas de mutaciones del VIH asociadas con la resistencia a los antirretrovirales. Esta detección de ácidos nucleicos escalable fundamentada en la técnica de edición del genoma, CRISPR lleva a los esfuerzos diagnósticos basados en pruebas específicas de alta prioridad hacia un testeo exhaustivo de grandes conjuntos de muestras, beneficiando enormemente a la salud pública
CARMEN
Los graves efectos de la pandemia por COVID-19 revelan nuestra vulnerabilidad ante enfermedades infecciosas emergentes. También pone de relieve la necesidad de contar con herramientas para detectar patógenos, tanto conocidos como de reciente aparición, que podrían amenazar la salud pública. Sin embargo, la diversidad genética de los posibles agentes, que incluyen virus, bacterias, hongos y protozoos, presenta una dificultad práctica. Los métodos moleculares que detectan los ácidos nucleicos son especialmente adecuados para esta tarea porque esos agentes infecciosos contienen ADN, ARN o ambos, lo que permite su reconocimiento e identificación. Los métodos de vigilancia viables para el seguimiento de las infecciones emergentes deben tener una amplia capacidad de detección, ser adecuados para un uso de alto rendimiento y tener un bajo costo por prueba. En relación a este importante aspecto, una reciente publicación de Cheri Ackerman y colaboradores del Departamento de Ingeniería Biológica del Massachusetts Institute of Technology describen un intento de cumplir estos requisitos utilizando una plataforma de detección diagnóstica que llamado CARMEN (del inglés, Combinatorial Arrayed Reactions for Multiplexed Evaluation of Nucleic acids) una tecnología que permite evaluar múltiples ácidos nucleicos mediante reacciones combinadas (Nature. 2020 Jun;582(7811):277-282).
CARMEN es una extensión de SHERLOCK, una plataforma de diagnóstico previamente desarrollada por algunos miembros del mismo equipo (DOI: 10.1126/science.aas8836) que se construyó en base a la herramienta de biotecnología CRISPR, la que puede ser utilizada para editar selectivamente los ácidos nucleicos. CRISPR se basa en un sistema de defensa bacteriano. Su uso como herramienta de laboratorio depende de un ARN "guía" (también llamado ARN CRISPR) presente en un complejo con una enzima Cas. Si el ARN guía se une a un blanco de ácido nucleico que es complementario a él en secuencia, la Cas se activa y rompe el blanco.
Algunas proteínas Cas cortan los ácidos nucleicos objetivo solo en un sitio específico relacionado con la secuencia guía. Sin embargo, la Cas13 se diferencia de otras proteínas Cas en que solo digiere ARN y no ADN, y ejerce su actividad de corte de ARN en cualquier ARN cercano que encuentre. Esta propiedad puede utilizarse para generar una señal que indique la presencia de una secuencia de interés.
Este principio subyace en SHERLOCK y CARMEN. El ARN de un reportero es cortado de una manera no específica de secuencia por Cas13 si es que esta enzima se activa a través del reconocimiento de una secuencia específica. Esta división genera una señal fluorescente al separar dos componentes unidos al ARN reportero: un quencher (apagador) de fluorescencia y una molécula fluorescente (figura 1). CARMEN conserva la detección sensible y específica lograda por SHERLOCK, y añade la capacidad de detección simultánea de múltiples objetivos de ácido nucleico. Esto hace que el flujo de trabajo sea compatible con una configuración miniaturizada de alto rendimiento que permite una rápida respuesta a un bajo costo por prueba.
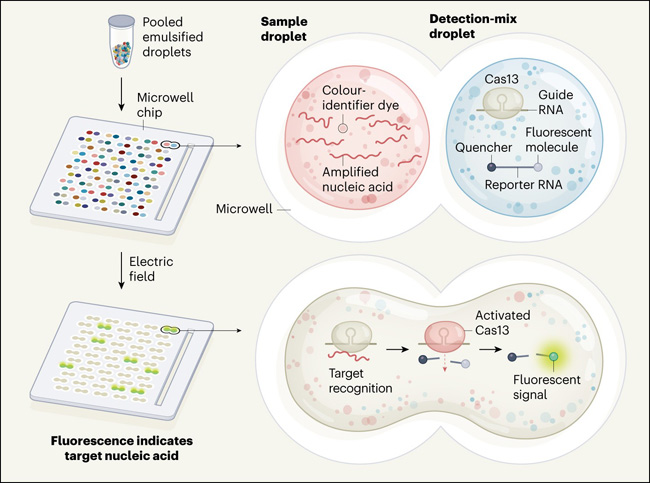
Figura 1. Un método para detectar la infección viral.
Cheri Ackerman y sus colegas presentan una plataforma tecnológica llamada CARMEN que puede identificar un rango de infecciones en una muestra dada, incluyendo la infección por el SARS-COv-2. Se necesitan dos categorías de gotas emulsionadas para esta plataforma: de muestra y gotitas que permitan la detección de un ácido nucleico específico. Cada tipo diferente de muestra o gota de detección contiene un identificador de color basado en un colorante único. Las gotitas mezcladas se cargan en un chip que contiene micropocillos que contienen dos gotitas aleatorias, y se registran los códigos de color. Si una muestra tiene secuencias virales de interés, estos ácidos nucleicos se amplifican cuando se preparan las gotas. Las gotas de la mezcla de detección contienen la enzima Cas13 en complejo con un ARN guía que permite el reconocimiento de una secuencia de ARN objetivo particular. También contienen un ARN reportero, unido a una molécula fluorescente que no emite señal debido a un quencher (apagador) unido al ARN. Cada par de gotas en un pocillo se fusiona cuando se aplica un campo eléctrico. Si el complejo Cas13 reconoce su ARN objetivo en el ácido nucleico amplificado, la Cas13 se activa y rompe el ARN reportero. Esto libera al apagador y genera una señal fluorescente, que revela una infección viral particular en la persona de la que procede la muestra.
Para detectar ácidos nucleicos específicos utilizando CARMEN, el proceso comienza con la amplificación de ácidos nucleicos virales objetivo (si están presentes) en un espécimen mediante métodos como la reacción en cadena de la polimerasa (PCR) o la amplificación por recombinasa y polimerasa (RPA). El ácido nucleico amplificado puede ser el producto de una reacción de amplificación específica dirigida a una sola secuencia viral, o puede ser el producto de reacciones combinadas utilizadas para amplificar potencialmente una gama de diferentes secuencias virales. La muestra de ARN amplificado recibe un código de color único mediante el uso de cuatro tintes fluorescentes mezclados en una proporción que confiere una de las 1.050 combinaciones de color posibles. A continuación, se añade aceite para generar gotas de un nanolitro emulsionadas. Los autores prepararon tales gotitas para todas las diferentes reacciones de amplificación llevadas a cabo. También generaron una serie de gotitas emulsionadas con códigos de colores únicos que contenían los componentes necesarios para detectar la presencia de secuencias virales específicas. Cada mezcla de detección estaba compuesta por un ARN reportero unido a un fluoróforo apagado y por Cas13 unido a un ARN guía necesario para detectar un objetivo viral.
Para ilustrar la posible aplicación de CARMEN para el análisis amplio de muestras virales, los autores muestran que la técnica podría detectar simultáneamente todos los 169 virus humanos para los que en ese momento se disponía de al menos 10 secuencias de su genoma. Los autores también demuestran que CARMEN permite la identificación exhaustiva de diferentes cepas de influenza a partir de muestras obtenidas de personas infectadas. Esto es importante, porque podría permitir la detección de un tipo de influenza de reciente aparición. La técnica también puede adaptarse para detectar una variante viral resultante de una mutación después de que se determine la secuencia de la variante. Ackerman y sus colegas informan que, cuando se utilizó en muestras de personas infectadas con VIH, pudo detectar seis mutaciones virales conocidas asociadas a la resistencia farmacológica.
Los autores ilustran la flexibilidad de CARMEN adaptando rápidamente el sistema para detectar el SARS-CoV-2, el coronavirus que causa la COVID-19. Los investigadores informan que la técnica distingue el SARS-CoV-2 de los demás coronavirus humanos, incluidos cuatro coronavirus estacionales y los responsables, respectivamente, del síndrome respiratorio agudo severo (SARS) y del síndrome respiratorio del Oriente Medio (MERS). Recientemente se ha informado de la detección rápida, sensible y específica del SARS-CoV-2 mediante un método que utiliza CRISPR y Cas12 (DOI: 10.1038/s41587-020-0513-4). Esta técnica tiene similitudes con el CARMEN, pero solo se puede detectar un tipo de virus.
Aunque CARMEN incorpora numerosas características deseables para la vigilancia de enfermedades infecciosas emergentes o la identificación de una infección viral, existen algunas preocupaciones. La primera es que el flujo de trabajo incluye la manipulación de ácido nucleico amplificado, por lo que existe el riesgo de contaminación. Tal vez una instrumentación automatizada apropiada podría reducir esta vulnerabilidad clave. En segundo lugar, ¿los efectos fuera de objetivo de la Cas13, posiblemente resultantes de la unión de ARN guía a objetivos incorrectos, conducirán a reacciones de detección inespecíficas? En tercer lugar, ¿será la generación y el análisis de imágenes de las nanogotas de estos chips lo suficientemente robusta en "condiciones de campo" en una gama de diferentes tipos de laboratorio, teniendo en cuenta la necesidad de un análisis sofisticado de microscopía de fluorescencia, y dado que los usuarios tendrán diferentes niveles de experiencia y conocimientos?
Por último, tal vez sea necesario modificar las secuencias utilizadas para amplificar el ARN y las secuencias de ARN guía utilizadas para lograr una sensibilidad y especificidad óptimas y tener en cuenta la variación del virus a lo largo del tiempo. Estos asuntos deben tenerse en cuenta, pero no disminuyen los logros del desarrollo de una nueva plataforma de diagnóstico diseñada en el contexto de una necesidad de vigilancia de enfermedades infecciosas emergentes a nivel mundial.
Fuente bibliográfica
CRISPR tool scales up to interrogate a huge line-up of viral suspects
Gregory A. Storch
School of Medicine, Washington University, St Louis, Missouri 63110, USA.
Nature. 2020 Jun;582(7811):188-189