Monitoreando medicamentos
El desarrollo de fármacos se enfrenta a múltiples retos que conducen a altos costos y largos ciclos de progreso de productos terapéuticos. Por su parte, el conocimiento sobre los procesos moleculares, celulares y fisiológicos han identificado un gran número de proteínas como posibles objetivos farmacológicos. Por lo tanto, se necesitan con urgencia métodos que promuevan el desarrollo acelerado de nuevos fármacos.
El efecto terapéutico de los medicamentos clínicamente disponibles se logra a través de la unión directa del fármaco a una o más proteínas diana. Esta unión se produce normalmente en un sitio funcional de la proteína, con un efecto activador o inhibidor. La modulación resultante de la actividad proteica en el contexto de las células y los tejidos conduce a respuestas moleculares, celulares y fisiológicas deseadas. La eficacia de un principio depende de forma crítica de la afinidad con su objetivo, y los efectos adversos son a menudo debido a la unión excesiva con proteínas dianas exponiendo a las células a la toxicidad.
Seguimiento a las drogas
Históricamente, el conocimiento de los medicamentos y sus objetivos se ha quedado muy por detrás de la administración medicamentosa a los pacientes, a menudo con resultados dañinos o incluso mortales. Varios avances sugieren que la identificación y validación de las interacciones fármaco-diana se pueden hacer con mayor facilidad, eficiencia y precisión. Una nueva estrategia de libre marcaje (que utiliza el fármaco nativo sin necesidad de identificación química o modificación) ideado por Daniel Martínez-Molina y colegas (Science 2013; 341:84-7), conocido como el ensayo de desplazamiento térmico celular (CETSA), es capaz de medir la unión fármaco-objetivo dentro de las células, facilitando el análisis de variables tales como el transporte, la activación, distribución tisular, especificidad de la diana y resistencia.
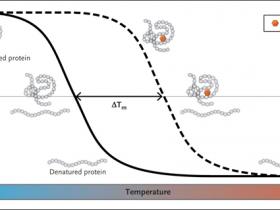
Cuando las proteínas en su conformación nativa son sometidas a condiciones de desnaturalización, incluyendo calor, pierden rápidamente sus estructuras de orden superior, lo que conduce al estado desplegado (figura 1). El desplegamiento de la proteína, se acompaña típicamente por cambios en las propiedades físicas (o biofísicas), que pueden ser disuadidos por la presencia de un ligando unido (o fármaco). Estas modificaciones incluyen el aumento en el tamaño de los agregados de la proteína o su afinidad por los colorantes polares que son la base para los ensayos térmicos de dispersión de luz y aquellos basados en fluorescencia - diferencial de dispersión de luz estática y fluorimetría diferencial de barrido, respectivamente - que son capaces de identificar ligandos (u otras condiciones) que aumentan la termoestabilidad de una proteína. Debido a que ambos métodos son independientes de la función proteica y son de alto rendimiento, han sido útiles en la biología estructural y la detección de drogas, pero se han limitado al análisis de proteínas puras.
En el fondo, el CETSA es una interpretación ingeniosa de ensayos de desnaturalización térmica (figura 1). Las alícuotas de lisados de células de mamífero cultivadas se tratan con un fármaco versus un control (o, alternativamente, se obtienen las células tratadas o tejidos de ratones tratados con un fármaco versus un control) y se calientan a varias temperaturas, lo que resulta en la formación, al aumentar la temperatura, de agregados de proteínas insolubles y desnaturalizados, que luego son convenientemente separados de sus homólogos solubles por centrifugación (después de la lisis celular). Los niveles de la proteína diana que quedan en la fracción soluble a cada temperatura se evalúan por medio de transferencia Western. La intensidad de la banda relativa de la proteína diana soluble restante se representa según la temperatura, y la temperatura de fusión de la proteína (en donde el 50% de la proteína se desnaturaliza) se deriva de la proteína libre frente a la que está unida al fármaco (figura 1), con la diferencia semicuantitativa correspondiente a la afinidad de la interacción molecular. La belleza de este método es que puede trabajar con mezclas complejas de proteínas (por ejemplo, células y tejidos enteros) en la vida real, incluyendo muchos escenarios clínicamente relevantes, sin que se requiera la generación de proteínas puras.
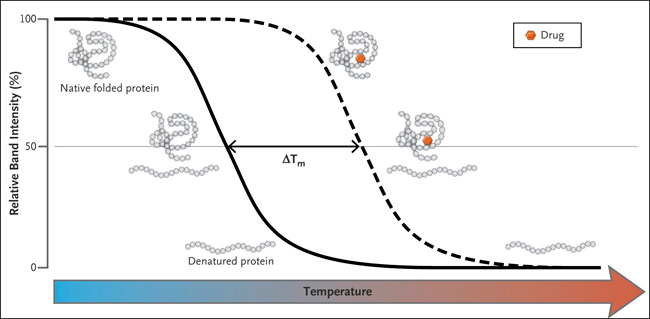
El equipo de Daniel Martínez-Molina utilizó el CETSA con un conjunto de medicamentos con objetivos clínicos conocidos, así como sus eficacias, analizaron sus efectos sobre la estabilidad de destino en células humanas cultivadas. Los resultados mostraron que en cada caso, la termoestabilidad de la proteína diana aumentaba cuando se añadía el fármaco de manera dosis-dependiente. Estos datos proporcionan la prueba que CETSA se puede aplicar generalmente a la supervisión de las interacciones fármaco-diana que se producen in vivo. Más allá de este análisis, el CETSA también puede ser útil para el descubrimiento de resultados desconocidos de los medicamentos (incluyendo efectos no deseados), de forma similar o complementaria a los métodos que explotan la estabilización inducida por ligando contra la proteólisis y la oxidación, respectivamente.
Esto demuestra que CETSA puede analizar la unión de fármacos de proteínas con grandes multidominios, que incluyen muchas de las metas terapéuticas en los seres humanos. Los posibles desafíos serían la agregación de proteínas multidominio por medio de dominios con diferentes estabilidades termodinámicas que se despliegan independientemente uno de otro. Sin embargo, el comportamiento de agregación de las proteínas puede ser modificado considerablemente en el entorno celular, en la acción de las chaperonas moleculares y aglomeración macromolecular, pudiendo permitir al CETSA realizar su trabajo.
El CETSA representa un método interesante para evaluar la eficacia del fármaco dentro de las células, en modelos animales, y potencialmente en pacientes. Dado que la mayoría de los principios fracasan en los ensayos clínicos y en la clínica debido a la toxicidad inesperada y a la falta de eficacia, un mayor desarrollo del CETSA y métodos relacionados, podrían promover el desarrollo de agentes terapéuticos seguros y efectivos.
Fuente bibliográfica
Tracking Drugs
Jing Huang, Ph.D.
Department of Molecular and Medical Pharmacology, David Geffen School of Medicine, and Molecular Biology Institute, University of California, Los Angeles, Los Angeles.
DOI: 10.1056/NEJMcibr1308868