Un revolucionario método para detectar patógenos
La detecciĂłn rápida, econĂłmica y sensible de ácidos nucleicos puede ayudar a la identificaciĂłn de patĂłgenos, a la genotipificaciĂłn y monitoreo de enfermedades en el punto de atenciĂłn. Dentro del conocido sistema molecular de ediciĂłn gĂ©nica CRISPR, existe una proteĂna efectora denominada Cas13a que exhibe un "efecto colateral" de actividad promiscua de ribonucleasa para el reconocimiento de una secuencia de ácido nucleico objetivo. Al combinar el efecto colateral de Cas13a con la amplificaciĂłn de secuencias se logra un preciso y rápido diagnĂłstico gracias a la detecciĂłn de molĂ©culas de ADN o ARN con sensibilidad attomolar y de especificidad de tan solo una base. La plataforma molecular basada en Cas13a, que fue denominada SHERLOCK (del inglĂ©s Specific High-Sensitivity Enzymatic Reporter UnLOCKing), además fue capaz de detectar cepas especĂficas del virus de Zika y Dengue, distinguir bacterias patĂłgenas, genotipificar ADN humano e identificar mutaciones en el ADN de tumores libres de cĂ©lulas. Una de las grandes virtudes es que los reactivos de SHERLOCK pueden liofilizarse para la independencia de la cadena frĂa y su almacenamiento a largo plazo. Pero quizás lo más relevante es su sencilla y potencial aplicaciĂłn en lugares de atenciĂłn primaria por personal de salud no familiarizado con laboratorio clĂnico, lo que representarĂa un gran avance para la rapidez diagnĂłstica de enfermedades infecciosas.
Detección de ácidos nucleicos
Durante los Ăşltimos 20 años, los grandes avances metodolĂłgicos como la reacciĂłn en cadena de la polimerasa (PCR) y la secuenciaciĂłn del ADN han transformado laboratorios de microbiologĂa y virologĂa clĂnica. Estos nuevos mĂ©todos permiten obtener resultados precisos y un diagnĂłstico rápido de una amplia gama de enfermedades infecciosas y facilitar el seguimiento de respuestas para el tratamiento de las infecciones, como las causadas por el virus de inmunodeficiencia humana (VIH) y citomegalovirus (CMV). Sin embargo, aĂşn existe una importante brecha en el arsenal de diagnĂłstico: la carencia de pruebas diagnĂłsticas rápidas, fiables, fáciles de usar y econĂłmicas que se pueden aplicar en el lugar de atenciĂłn. Desde hace años se viene demandando este tipo de proyectos de pruebas en áreas con recursos limitados, pero tales pruebas pueden tener importancia en un amplio rango de análisis, proporcionando resultados que pueden afectar decisiones clĂnicas en tiempo real.Â
Para ello, Jonathan Gootenberg y colaboradores (doi: 10.1126/science.aam9321) reprogramaron una endonucleasa que se asocia con CRISPR (repeticiones palindrĂłmicas cortas agrupadas y regularmente interespaciadas) en el ADN de procariotas (estas secuencias son parte del sistema inmunitario adaptativo de los procariotas) para lograr una sensibilidad analĂtica a nivel de molĂ©cula individual, para una rápida detecciĂłn de ácidos nucleicos. Los investigadores explotaron una actividad de la enzima Cas13a llamado "actividad ARNasa promiscua". Una vez que la enzima rompe un objetivo de ARN (como una secuencia especĂfica de virus a la que se guĂa especĂficamente por un complemento de ARN), puede unirse y degradar a otros fragmentos de ARN, como los ligados a fluorĂłforos, los que sirven como reporteros (figura 1). Combinando la detecciĂłn basada en Cas13a con la amplificaciĂłn isotĂ©rmica del ADN y del ARN objetivos en un solo tubo de reacciĂłn, se logra una rápida visualizaciĂłn y detecciĂłn en tiempo real de objetivos, incluso a bajas concentraciones, y la diferenciaciĂłn de productos especĂficos en ARN o ADN.
Para determinar si este mĂ©todo proporciona la sensibilidad y especificidad requeridas para el diagnĂłstico de enfermedades infecciosas, los autores diseñaron lentivirus que contenĂan fragmentos del genoma del virus Zika (ZIKV) y del virus del dengue (DENV). El sistema detectĂł secuencias ZIKV en concentraciones tan bajas como 2 attomolar (aM) y distinguiĂł diferencialmente a ambos tipos de patĂłgenos, una caracterĂstica importante para aplicaciones clĂnicas (figura 2). Los autores entonces exploraron el uso potencial de este mĂ©todo en campo liofilizando los reactivos y posteriormente rehidratándolos.
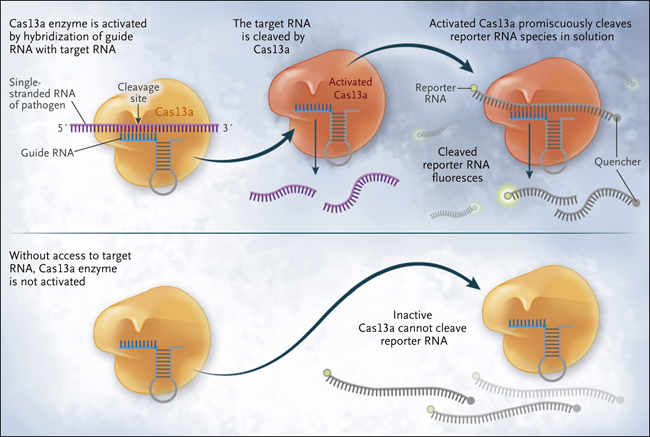
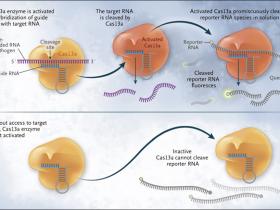
Figura 1. El mecanismo de SHERLOCK.
La reacción SHERLOCK combina una preamplificación del ADN mediante amplificación por polimerasa recombinasa (RPA) o una preamplificación de ARN con RPA con transcripción inversa con una detección posterior dirigida por Cas13a. Durante la preamplificación, se añaden los promotores de la ARN polimerasa T7 para permitir la transcripción del ADN amplificado a RNA. Este ARN puede ser detectado a través de incubación con Cas13a, ARN CRISPR complementarios y sensores fluorescentes de ARN. Al unirse a una secuencia de ARN objetivo, la enzima Cas13a se activa y rompe promiscuamente otras especies de ARN, fenómeno conocido como efecto colateral. Los sensores de ARN con reporteros fluorescentes en el extremo 5' y una molécula que reduce la fluorescencia (quencher) en el extremo 3' son divididas por Cas13a, generando una señal fluorescente. En ausencia de la activación de Cas13a, no ocurre la escisión del ARN reportero ni la generación de fluorescencia. La combinación de pasos de amplificación y este sistema de detección permite la identificación con especificidad de una sola base en 1 a 2 horas.
El sistema basado en papel para la detecciĂłn, funcionĂł pero resultĂł en una reducciĂłn general de señal fluorescente para ZIKV y un ruido más alto para DENV, de forma que la señal de detecciĂłn se redujo en aproximadamente 1 log10 (20 aM). El rendimiento del mĂ©todo de detecciĂłn fue tambiĂ©n evaluado con el uso de muestras de suero y orina de cuatro pacientes infectados con ZIKV; el test detectĂł el virus en las cuatro muestras clĂnicas, con concentraciones de ARN que iban desde 8,25Ă—10e5 copias por mililitro (copias /mL) a 1,25Ă—10e3 (copias /mL) verificado por medio de PCR cuantitativo en tiempo real.Los autores tambiĂ©n mostraron cĂłmo el mĂ©todo, SHERLOCK (del inglĂ©s specific high-sensitivity enzymatic reporter unlocking), identifica patĂłgenos bacterianos al dirigirse al gen de ARNr 16S, particularmente en la regiĂłn V3. Comenzando desde una colonia aislada, SHERLOCK pudo detectar Escherichia coli y Pseudomonas aeruginosa y distinguir estas especies de Klebsiella pneumoniae, Mycobacterium tuberculosis, y Staphylococcus aureus. Además, SHERLOCK puede diferenciar fácilmente los aislados clĂnicos de K. pneumoniae que poseen genes de resistencia a carbapenemasa o a beta lactámicos. Finalmente, la especificidad de SHERLOCK fue mejorada mediante la introducciĂłn de cambios sintĂ©ticos en la guĂa de ARN que condujo a la generaciĂłn de una o más bases desapareadas cuando se hibridan con el ARN objetivo.
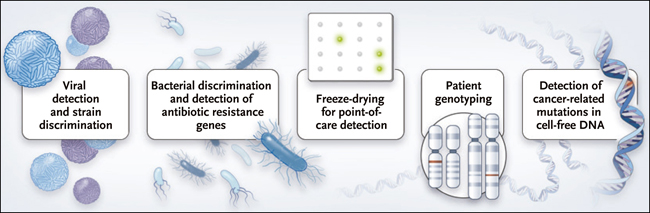
Figura 2. Versatilidad en la aplicaciĂłn.
Los autores mostraron la aplicación de SHERLOCK en varios escenarios. Entre ellos se incluye la detección de una sola molécula del virus de Zika y la distinción de cepas estrechamente relacionadas. Además lograron discriminar diferentes especies de bacterias y detectar genes de resistencia a antibióticos; la determinación rápida del genotipo del paciente y detección de mutaciones relacionadas con el cáncer en el ADN libre de células.
Este ensayo modificado permitiĂł la discriminaciĂłn de los blancos que difieren en sĂłlo un solo par de bases y distinguiĂł exitosamente las cepas africanas y americanas de ZIKV, diferentes serotipos de DENV, cinco alelos de relevancia clĂnica desde la saliva, y varias mutaciones relacionadas con el cáncer en suspensiones de ADN libre de cĂ©lulas. Esto Ăşltimo representa una de las aplicaciones más potentes de SHERLOCK. TambiĂ©n tiene varias caracterĂsticas importantes que lo hacen factible para su uso en el punto de atenciĂłn, dado la velocidad con que se realiza un ensayo en papel pueden diseñarse y sintetizarse (sĂłlo en pocos dĂas), el coste estimado de los reactivos y materiales (menos de $1 por prueba), la estabilidad de los reactivos liofilizados, y la velocidad con que se puede realizar el ensayo (dentro de 1 a 2 horas).
Aunque los resultados descritos son alentadores, se necesita una evaluaciĂłn más rigurosa de las caracterĂsticas de rendimiento del sistema SHERLOCK para cada uno de las aplicaciones. Además, el proceso de comercializaciĂłn y la introducciĂłn de nuevas tecnologĂas en la práctica clĂnica es un reto. La prueba tendrĂa que tener caracterĂsticas de rendimiento que se asemejan a los actuales mĂ©todos de laboratorio utilizados en el análisis clĂnico para muestras de pacientes y, ser diseñada con la simplicidad necesaria para su uso en el punto de atenciĂłn por personal clĂnico ajeno al laboratorio. En el pasado, algunos mĂ©todos diagnĂłsticos tradicionales, como el cultivo viral y las pruebas de detecciĂłn rápida de antĂgenos, fueron tan limitadas en su rendimiento general que las nuevas tecnologĂas moleculares se incorporaron rápidamente al uso clĂnico.Â
Sin embargo, actualmente las pruebas deben demostrar que mejorarán la calidad clĂnica costo-eficacia para obtener la certificaciĂłn y financiamiento para la realizaciĂłn de estudios pertinentes. Aunque hay una clara necesidad de pruebas rápidas y econĂłmicas en el punto de atenciĂłn, y aunque el entusiasmo por la posibilidad que la tecnologĂa SHERLOCK pueda, tal como ocurriĂł con la PCR, representar un gran avance en tĂ©rminos de velocidad y la precisiĂłn de detecciĂłn de muchos patĂłgenos microbianos, aĂşn se espera ansiosamente por estudios más detallados para determinar si este innovador mĂ©todo es capaz de llenar el gap de diagnĂłstico.
Fuente bibliográfica
A CRISPR Way to Diagnose Infectious Diseases
Angela M. Caliendo, M.D., Ph.D., and Richard L. Hodinka, Ph.D.
Department of Medicine, Warren Alpert Medical School of Brown University, Providence.
DOI: 10.1056/NEJMcibr1704902