Defensa microbiana para descubrir antivirales
Los beneficios de las nuevas herramientas para la edición génica en medicina y en ciencias biomédicas representan promisorias oportunidades para combatir enfermedades. Estos novedosos métodos utilizan la maquinaria programable de corte del ADN de un sistema inmunológico bacteriano (CRISPR- Cas9, del inglés clustered regularly interspaced short palindromic repeats), que puede ser adaptado para casi cualquier tipo de célula. Con el uso de guías consistentes en moléculas de ARN que orientan a la enzima Cas9 hacia una región específica en el genoma, se puede lograr la inactivación o edición de diferentes genes en células de cultivo o en modelos animales. Incluso, tal vez a futuro - si las limitaciones técnicas y éticas son superadas- este método será utilizado para sanar patologías humanas.
Edición génica y proteínas
El método CRISPR trabaja con tal eficiencia que es posible (con las advertencias que se detallan a continuación) evaluar simultáneamente el papel de cada gen humano en un proceso biológico. Park y colaboradores realizaron exactamente esto en el contexto del virus de la inmunodeficiencia humana tipo 1 (VIH-1). Tomaron una línea de células T que normalmente serían susceptibles a la replicación viral, diseñadas para expresar Cas9, e introdujeron muchos miles de guías CRISPR. Cada guía era capaz de generar un knockout de un gen humano individual. (Nat Genet 2016; 49: 193-203). Luego, tras desafiar poblaciones de células que contienen CRISPR-Cas9 con VIH-1, los investigadores seleccionaron las células que resistieron a la infección y determinaron cual de las guías de CRISPR (y por lo tanto qué genes knockouts) conferían tal resistencia a VIH-1 (Figura 1).
Los resultados evidenciaron que cinco knockouts genéticos confirieron una resistencia robusta al VIH-1. Cuatro de estos genes estaban directa o indirectamente involucrados en el proceso de entrada viral en las células. Dos genes (CD4 y CCR5) codifican un para un receptor y coreceptor para el virus, y otros dos (TPST2 y SLC35B2) codifican una enzima de sulfatación y un transportador de donantes de sulfato, respectivamente. Estos hallazgos tienen sentido, dado que la sulfatación de CCR5 es fundamental para la entrada eficiente del VIH-1 a las células. El quinto gen (ALCAM) codifica una molécula de adhesión celular que hace que las células se agrupen en el cultivo, lo que acelera la replicación viral facilitando la transferencia de viriones de una célula infectada a una población celuar vecina aún no infectada. Los niveles de expresión de ALCAM no son elevados en los linfocitos T y pueden no ser importantes bajo condiciones in vivo. Sin embargo, la investigación de Park mostró que otras moléculas de adhesión son relevantes para la propagación del VIH-1 en células primarias in vitro y podrían contribuir plausiblemente a la propagación viral in vivo.
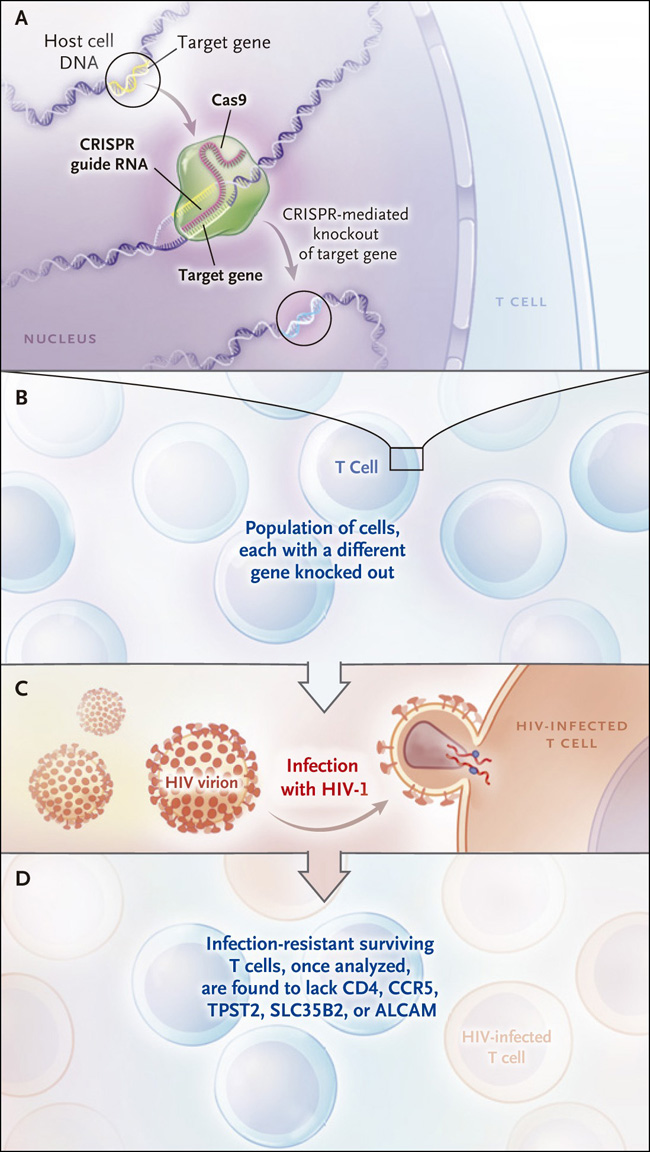
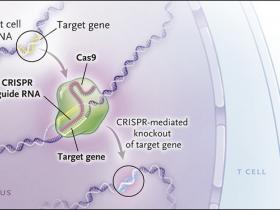
Figura 1: El método CRISPR para identificar proteínas esenciales para la infección por el VIH-1.
Se disenó una línea de células T para expresar Cas9 (una enzima capaz de cortar el ADN) y un ARN guía CRISPR, que permite localizar a Cas9 en un lugar específico del genoma humano. Cas9 genera rupturas e inactivación del gen blanco del sistema CRISPR. Cada célula contiene un ARN CRISPR diferente y por lo tanto, diferentes genes knockout. Cada gen humano bloqueado que es compatible con la viabilidad celular está representado en la población (paneles A y B). La población de células fue desafiada con el VIH de tipo 1 (VIH - 1) (Panel C). Las células no infectadas que sobrevivieron carecían de moléculas necesarias para la entrada de VIH-1 (CD4, CCR5, TPST2 o SLC35B2) o para la adhesión de células entre sí (ALCAM) (panel D).
Es sorprendente que mediante el uso del enfoque CRISPR solo se reveló un número muy pequeño de genes necesarios para la infección. Las investigaciones realizadas utilizando una tecnología genómica funcional (ARN interferentes) sugirió que cientos de genes del hospedero fueron importantes para la infección por VIH-1. Hay algunas razones para esta discrepancia. El diseño de cribado utilizado por Park permitió la identificación de genes que son importantes en etapas tempranas del ciclo replicativo del virus, pero no en las más complejas ni en etapas tardías dependientes del hospedero. Además, a diferencia del ARN interferentes, los knockouts CRISPR deben ser compatibles con la viabilidad celular a más largo plazo.
Como Park y sus colegas señalan, muy pocos de los genes identificados como críticos para la replicación del VIH-1 han resistido los rigores de la investigación posterior como para establcerse como esenciales para la infección viral. Muchos de los genes identificados en el método de ARN interferente probablemente ejercen efectos indirectos en la replicación del VIH-1 a través de perturbaciones en la fisiología celular, y sus knockouts no han superado el riguroso obstáculo inherente a la viabilidad del cribado de CRISPR. De hecho, CRISPR y los métodos de ARN interferente no identifican muchos genes bien establecidos para ser cruciales para la replicación del VIH-1, probablemente porque la mayoría de los productos génicos del hospedero son explotados por el VIH-1 y son también necesarios para la supervivencia celular. Si el objetivo es obtener una descripción molecular completa de la replicación del VIH-1, entonces la debilidad del los métodos de este tipo es que no identifican productos de genes esenciales. Por otro lado, si el objetivo es identificar proteínas celulares que pueden ser enfoque de nuevos medicamentos, el método basado en CRISPR ofrece ventajas notables.
¿Cómo podrían ser explotados estos hallazgos para mejorar el tratamiento de la infección por VIH-1? De hecho, ya lo han realizado. Maraviroc, una pequeña molécula de unión a CCR5, es un inhibidor clínicamente eficaz y que ha sido aprobado para la infección por VIH-1. Incluso, es el único inhibidor aprobado contra VIH-1 que se dirige a un hospedero en lugar de hacerlo hacia una proteína viral. El hecho que las principales conclusiones de Park y sus colaboradores ya han sido explotadas terapéuticamente sirve para ilustrar el poder de una identificación rápida de los productos génicos celulares más prometedores que pueden ser blanco de nuevas terapias.
Fuente bibliográfica
Repurposing a Bacterial Immune System to Discover Antiviral Targets
Paul Bieniasz, Ph.D.
Laboratory of Retrovirology, Rockefeller University, New York.
DOI: 10.1056/NEJMcibr1616057
Temas Relacionados